קובץ:Coronavirus. SARS-CoV-2.png
לקובץ המקורי (2,048 × 2,048 פיקסלים, גודל הקובץ: 4.54 מגה־בייטים, סוג MIME: image/png)
זהו קובץ מתוך ויקישיתוף וניתן להשתמש בו גם במיזמים אחרים. תיאורו בדף תיאור הקובץ שלו מוצג למטה.
תקציר
| תיאורCoronavirus. SARS-CoV-2.png |
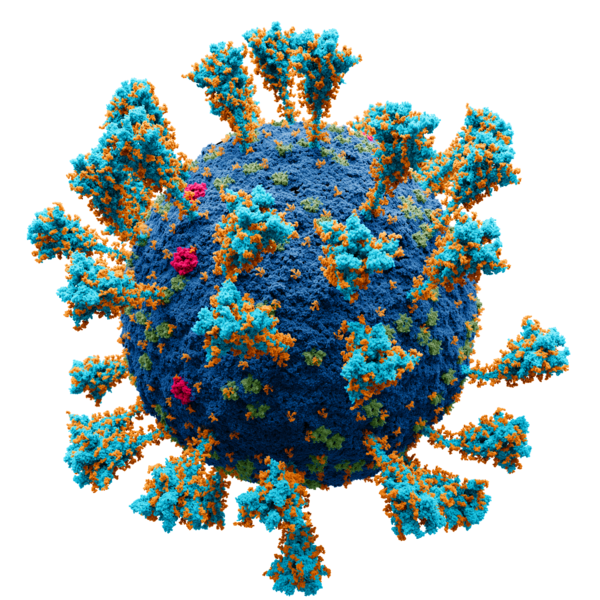
Deutsch: Wissenschaftlich genaues Atommodell der äußeren Struktur des SARS Coronavirus 2 (SARS-CoV-2), einem Stamm (genetische Variante) des Coronavirus, der die Coronavirus-Krankheit (COVID-19) verursachte und erstmals im Dezember 2019 in Wuhan, China, identifiziert wurde.
Jeder einzelne Ort (amorpher Fleck) ist ein Atom von: English: Scientifically accurate atomic model of the external structure of the Severe Acute Respiratory Syndrome CoronaVirus 2 (SARS-CoV-2), a strain (genetic variant) of the coronavirus that caused coronavirus disease (COVID-19), first identified in Wuhan, China, during December 2019
Each separate locus (amorphous blob) is a molecule of: Español: Modelo atómico de la estructura externa del SARS-CoV-2. Cada "bola" es un átomo.
Русский: Научно достоверная атомарная модель внешней структуры коронавируса (SARS-CoV-2). Каждый "шарик" — атом. Опубликовано на N+1.
Научный консультант:
Protein Data Bank: 2mls, 6y3y, 5x29, 6yyt Charmm-gui: 6vsb_1_1_1_S309
Проект был сделан в соответствии с научными источниками:
Первичные источники:
|
| תאריך יצירה | |
| מקור |
נוצר על־ידי מעלה היצירה. Scientific consultants:
|
| יוצר | Alexey Solodovnikov (Idea, Producer, CG, Editor), Valeria Arkhipova (Scientific Сonsultant) |
| אישורים והיתרים (שימוש חוזר בקובץ זה) |
Published by N+1 a popular science online publication of Russia (https://nplus1.ru/), protein models are derivative works of the free license site (freely available for both non-commercial and commercial use) |
| גרסאות אחרות |
|
Sources
Primary sources:
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7489918/
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7098027/
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7605623/
The following structures from open sources were used in the work, Protein Data Bank (https://www.rcsb.org):
- 2mls (membrane bilayer complex with matrix metalloproteinase-12 at its beta-face) Koppisetti, R.K., Fulcher, Y.G., Prior, S.H., Lenoir, M., Overduin, M., Van Doren, S.R. 2014
- 6y3y (human coronavirus HKU1 haemagglutinin-esterase) Hurdiss, D.L., Drulyte, I., Pronker, M.F. 2020
- 5x29 (NMR structure of the SARS Coronavirus E protein pentameric ion channel) Torres, J., Surya, W., Li, Y. 2017
- 6yyt (Structure of replicating SARS-CoV-2 polymerase) Hillen, H.S., Kokic, G., Farnung, L., Dienemann, C., Tegunov, D., Cramer, P. 2020
- Charmm-gui: 6vsb_1_1_1_S309
Additional sources:
- Surya, W., Li, Y., Torres, J. Structural model of the SARS coronavirus E channel in LMPG micelles // Biochim. Biophys. Acta - Biomembr. – 2018. – Vol. 1860. – N. 6. – P. 1309–1317.
- Koppisetti, R. K., Fulcher, Y. G., Jurkevich, A., Prior, S. H., Xu, J., Lenoir, M., Overduin, M., Van Doren, S. R. Ambidextrous binding of cell and membrane bilayers by soluble matrix metalloproteinase-12 // Nat. Commun. – 2014. – Vol. 5. – P. 1–14.
- Hillen, H. S., Kokic, G., Farnung, L., Dienemann, C., Tegunov, D., Cramer, P. Structure of replicating SARS-CoV-2 polymerase // Nature. – 2020. – Vol. 584. – N. 7819. – P. 154–156.
- Harris, L. J., Larson, S. B., Hasel, K. W., McPherson, A. Refined structure of an intact IgG2a monoclonal antibody // Biochemistry. – 1997. – Vol. 36. – N. 7. – P. 1581–1597.
- Noreng, S., Bharadwaj, A., Posert, R., Yoshioka, C., Baconguis, I. Structure of the human epithelial sodium channel by cryo-electron microscopy // Elife. – 2018. – Vol. 7. – P. 1–23.
- Almond, A., DeAngelis, P. L., Blundell, C. D. Hyaluronan: The Local Solution Conformation Determined by NMR and Computer Modeling is Close to a Contracted Left-handed 4-Fold Helix // J. Mol. Biol. – 2006. – Vol. 358. – N. 5. – P. 1256–1269.
- Hurdiss, D. L., Drulyte, I., Lang, Y., Shamorkina, T. M., Pronker, M. F., van Kuppeveld, F. J. M., Snijder, J., de Groot, R. J. Cryo-EM structure of coronavirus-HKU1 haemagglutinin esterase reveals architectural changes arising from prolonged circulation in humans // Nat. Commun. – 2020. – Vol. 11. – N. 1. – P. 1–10.
- Yan, Renhong, Yuanyuan Zhang, Yaning Li, Lu Xia, Yingying Guo, Q. Z. Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2 // Science (80-. ). – 2020. – Vol. 3. – N. 3. – P. 1–8.
- Javitt, G., Khmelnitsky, L., Albert, L., Bigman, L. S., Elad, N., Morgenstern, D., Ilani, T., Levy, Y., Diskin, R., Fass, D. Assembly Mechanism of Mucin and von Willebrand Factor Polymers // Cell. – 2020. – Vol. 183. – N. 3. – P. 717-729.e16.
- Daniel Wrapp, Nianshuang Wang, Kizzmekia S. Corbett, Jory A. Goldsmith, Ching-Lin Hsieh, Olubukola Abiona, B. S. G., McLellan, and J. S. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation // Science (80-. ). – 2020. – Vol. 21. – N. 1. – P. 1–9.
- Wang, M. Y., Zhao, R., Gao, L. J., Gao, X. F., Wang, D. P., Cao, J. M. SARS-CoV-2: Structure, Biology, and Structure-Based Therapeutics Development // Front. Cell. Infect. Microbiol. – 2020. – Vol. 10. – N. November. – P. 1–17. (https://pubmed.ncbi.nlm.nih.gov/33324574/)
- Yao, H., Song, Y., Chen, Y., Wu, N., Xu, J., Sun, C., Zhang, J., Weng, T., Zhang, Z., Wu, Z., Cheng, L., Shi, D., Lu, X., Lei, J., Crispin, M., Shi, Y., Li, L., Li, S. Molecular Architecture of the SARS-CoV-2 Virus // Cell. – 2020. – Vol. 183. – N. 3. – P. 730-738.e13.
- Oostra, M., de Haan, C. A. M., de Groot, R. J., Rottier, P. J. M. Glycosylation of the Severe Acute Respiratory Syndrome Coronavirus Triple-Spanning Membrane Proteins 3a and M // J. Virol. – 2006. – Vol. 80. – N. 5. – P. 2326–2336. (https://europepmc.org/article/MED/16474139)
- B.W. Neuman, M. J. B. Supramolecular Architecture of the Coronavirus Particle // Adv. Virus Res. – 2020. – Vol. 96. – P. 1–27 (https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7112365/, https://europepmc.org/article/PMC/1563832)
- Neuman, B. W., Kiss, G., Kunding, A. H., Bhella, D., Baksh, M. F., Connelly, S., Droese, B., Klaus, J. P., Makino, S., Sawicki, S. G., Siddell, S. G., Stamou, D. G., Wilson, I. A., Kuhn, P., Buchmeier, M. J. A structural analysis of M protein in coronavirus assembly and morphology // J. Struct. Biol. – 2011. – Vol. 174. – N. 1. – P. 11–22. (https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4486061/)
- Yu, A., Pak, A. J., He, P., Monje-Galvan, V., Casalino, L., Gaieb, Z., Dommer, A. C., Amaro, R. E., Voth, G. A. A multiscale coarse-grained model of the SARS-CoV-2 virion // Biophys. J. – 2021. – Vol. 120. – N. 6. – P. 1097–1104 (https://europepmc.org/article/PMC/PMC7695975, https://search.bvsalud.org/global-literature-on-novel-coronavirus-2019-ncov/resource/en/covidwho-947143)
- Yao, H., Song, Y., Chen, Y., Wu, N., Xu, J., Sun, C., Zhang, J., Weng, T., Zhang, Z., Wu, Z., Cheng, L., Shi, D., Lu, X., Lei, J., Crispin, M., Shi, Y., Li, L., Li, S. Molecular architecture of the SARS-CoV-2 virus // Cell. – 2020. – Vol. 183. – N. 3. – P. 730–738 (https://www.sciencedirect.com/science/article/pii/S0092867420311594)
- Choi, Y. K., Cao, Y., Frank, M., Woo, H., Park, S. J., Yeom, M. S., Croll, T. I., Seok, C., Im, W. Structure, Dynamics, Receptor Binding, and Antibody Binding of the Fully Glycosylated Full-Length SARS-CoV-2 Spike Protein in a Viral Membrane // J. Chem. Theory Comput. – 2021. – Vol. 17. – N. 4. – P. 2479–2487 (https://www.researchgate.net/publication/349986293_Structure_Dynamics_Receptor_Binding_and_Antibody_Binding_of_the_Fully_Glycosylated_Full-Length_SARS-CoV-2_Spike_Protein_in_a_Viral_Membrane)
רישיון
- יש לך חופש:
- לשתף – להעתיק, להפיץ ולהעביר את העבודה
- לערבב בין עבודות – להתאים את העבודה
- תחת התנאים הבאים:
- ייחוס – יש לתת ייחוס הולם, לתת קישור לרישיון, ולציין אם נעשו שינויים. אפשר לעשות את זה בכל צורה סבירה, אבל לא בשום צורה שמשתמע ממנה שמעניק הרישיון תומך בך או בשימוש שלך.
- שיתוף זהה – יצירת רמיקס, שינוי או בנייה על סמך החומר הזה, תטיל עליך חובה להפיץ את התרומות שלך לפי תנאי רישיון זהה או תואם למקור.
הערכה
|
כיתובים
פריטים שמוצגים בקובץ הזה
מוצג
ערך כלשהו ללא פריט ויקינתונים
4 במאי 2021
Commons quality assessment אנגלית
image/png
4,765,767 בית
2,048 פיקסל
2,048 פיקסל
היסטוריית הקובץ
ניתן ללחוץ על תאריך/שעה כדי לראות את הקובץ כפי שנראה באותו זמן.
| תאריך/שעה | תמונה ממוזערת | ממדים | משתמש | הערה | |
|---|---|---|---|---|---|
| נוכחית | 22:17, 9 בינואר 2022 |  | 2,048 × 2,048 (4.54 מגה־בייטים) | Jul059 | Lossless file size reduction |
שימוש בקובץ
הדף הבא משתמש בקובץ זה:


 כניסה
כניסה  עקבו אחרינו בפייסבוק
עקבו אחרינו בפייסבוק 


